目前很多融合基因都是基于高通量测序检测到的。如果要涉及到高通量测序的话,那就可以使用TCGA的数据来分析融合基因。同时TCGA又具有多组学的数据。所以也就可以分析融合基因和多组学的关系。因此也就有了下面这个融合基因多组学分析数据库:
FPIA: A webserver for Fusion Profiling Interactive Analysis:
http://bioinfo-sysu.com/fpia/public/fusion/
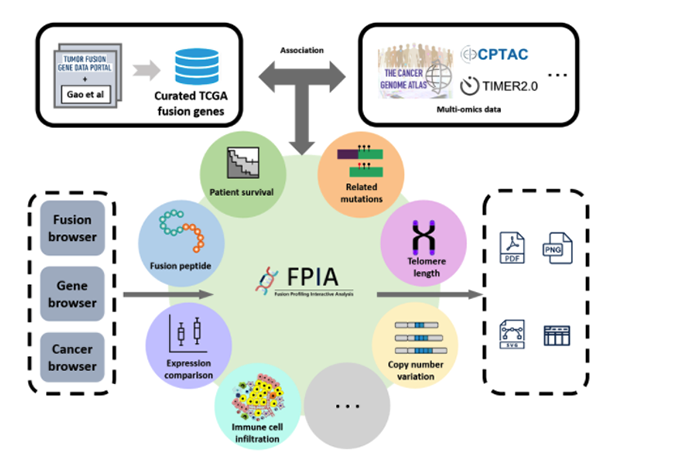
背景数据集介绍
FPIA分析的主要数据来源就是TCGA数据库。作者使用TCGA多组学的数据。首先鉴定了TCGA每个癌种有多少个融合基因事件。同时基于TCGA多组学的数据(突变,拷贝数,免疫浸润,生存信息,蛋白MS)来分析融合基因和这些多组学的关系。
数据库使用
在使用数据库进行多组学融合基因分析之前,首先还是需要进行融合基因的检索的。查看一下是否有我们想要检索的融合基因。在FPIA当中提供了三种查找融合基因信息的方式:
1)某一个基因检索;2)具体融合基因检索;3)某一个肿瘤检索
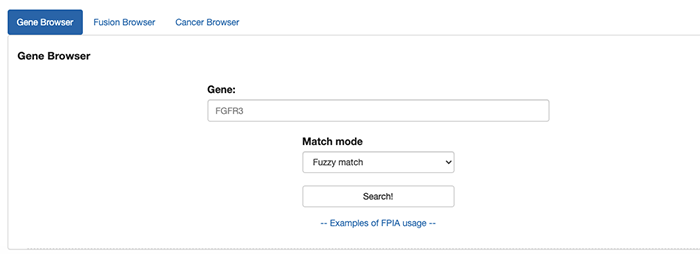
例如我们这里查询:FGFR3有关的融合基因。就可以在基因检索当中输入:FGFR3.。经过检索就可以得到和这个基因有关的融合基因信息。其中包括这个融合基因的类型,检测的癌种,基因的类型等等
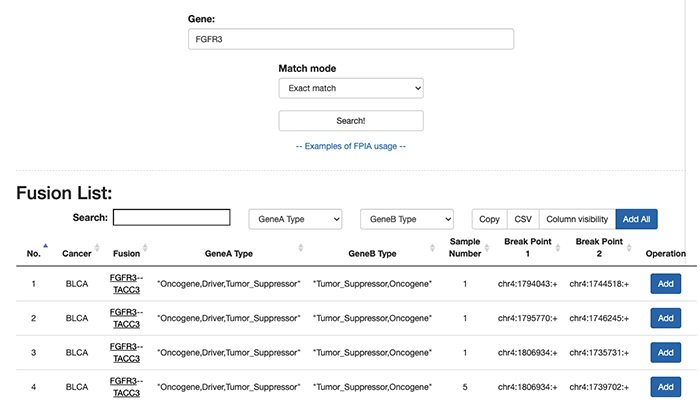
进一步可以选择想要分析的融合基因类型进行分析。比如我们可以选择Add all来分析所有和FGFR有关的融合基因,之后就可以对这些融合基因进行分析了
其中涉及到的分析包括:1)基因表达;2)肿瘤纯度;3)突变;4)拷贝数变异;5)蛋白表达;6)免疫浸润;7)细胞干性;8)端粒长度;9)微卫星不稳定性;10)生存分析以及11)新的肽段
比如在基因表达当中,就可以观察某一个基因表达在基因融合不融合的情况下是否存在差异。结果是以箱式的类型来进行展示。
再比如在观察融合基因与否和肿瘤预后是否有关系。
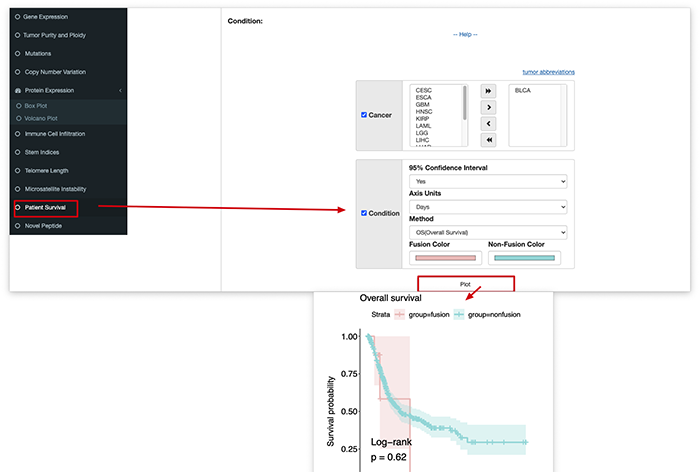
至于其他的,有兴趣的可以去分析一下看看。
以上就是关于FPIA的主要使用方法。主要还是基于TCGA的多组学数据来分析融合基因和不同组学的关系。如果要研究融合基因的可以尝试的进行多组学分析试一试













